PythonとmatplotlibでDNAの立体図(pdb)を表示する
Pythonを介したmatplotlibの使用により、pdbファイルとして得たDNAの立体データをxyz座標のみのデータに変換し、ウィンドウに表示させます。
- matplotlib …Pythonのグラフ描画ライブラリ(公式サイト)
- pdbファイル …PDB(Protein Data Bank)が公開している、X線やNMRを用いた結晶構造解析の結果を記したデータファイル(公式サイト)
今回用いる方法では原子半径を考慮せずに全て均一なサイズの点として表現するため、あくまで原子中心間の距離関係がわかるものとしてお取り扱いください。
用いるデータの取り扱いなどは「[PythonとPlotlyでDNAの立体図をブラウザ上で表示する]」と同様です。データの取得や加工につきましてはそちらをご参照ください。
関連記事
index
コード全体
動作環境:
- python 3.6.8
- matplotlib 3.0.3
- windows10 64bit
| input | output |
|---|---|
| input.pdb | ウィンドウに表示 |
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
import numpy as np
data = open("input.pdb", "r")
with open('pdb.txt', 'w') as f:
lines = data.readlines()
line_list = []
for i in range(len(lines)):
if 'ATOM' in lines[i][0:4]:
line = lines[i].replace(lines[i][0:31], '').replace(lines[i][54:80], '')
line_list.append(line)
for i in range(len(line_list)):
f.write(line_list[i])
data.close()
pts = np.loadtxt('pdb.txt')
x, y, z = pts.T
fig = plt.figure()
ax = fig.gca(projection='3d')
ax.set_xlim([0, 30])
ax.set_ylim([0, 30])
ax.set_zlim([0, 30])
ax.scatter(x, y, z, s=100, c=z)
ax.view_init(elev=0, azim=0)
ax.set_xticks([])
ax.set_yticks([])
ax.set_zticks([])
ax.xaxis.set_pane_color((1,1,1,0))
ax.yaxis.set_pane_color((1,1,1,0))
ax.zaxis.set_pane_color((1,1,1,0))
ax.xaxis._axinfo["grid"]['color'] = (1,1,1,0)
ax.yaxis._axinfo["grid"]['color'] = (1,1,1,0)
ax.zaxis._axinfo["grid"]['color'] = (1,1,1,0)
ax._axis3don = False
plt.show()
重要な箇所を以下に示します。
データの読み込みと変換
data = open("input.pdb", "r")
with open('pdb.txt', 'w') as f:
lines = data.readlines()
line_list = []
for i in range(len(lines)):
if 'ATOM' in lines[i][0:4]:
line = lines[i].replace(lines[i][0:31], '').replace(lines[i][54:80], '')
line_list.append(line)
for i in range(len(line_list)):
f.write(line_list[i])
data.close()
pts = np.loadtxt('pdb.txt')
x, y, z = pts.T
fig = plt.figure()
ax = fig.gca(projection='3d')
すでにinput.pdbは取得しているものとします。まだ取得していない場合は、こちらの手順にしたがって取得してください。今回はB-DNAのデータを取得しています。
pdbファイルを原子のxyz座標のみに変換したデータであるpdb.txtファイルをnp.loadtxtで読み込みます。読み込まれたデータは、1列目がx、2列目がy、3列目がzとして読み込まれます。
その後、グラフとして読み込んでいきます。
グラフパラメータの指定
ax.set_xlim([0, 30])
ax.set_ylim([0, 30])
ax.set_zlim([0, 30])
ax.scatter(x, y, z, s=100, c=z)
ax.view_init(elev=0, azim=0)
ax.set_xticks([])
ax.set_yticks([])
ax.set_zticks([])
ax.xaxis.set_pane_color((1,1,1,0))
ax.yaxis.set_pane_color((1,1,1,0))
ax.zaxis.set_pane_color((1,1,1,0))
ax.xaxis._axinfo["grid"]['color'] = (1,1,1,0)
ax.yaxis._axinfo["grid"]['color'] = (1,1,1,0)
ax.zaxis._axinfo["grid"]['color'] = (1,1,1,0)
ax._axis3don = False
各種グラフパラメータを変更していきます。以下に変更した箇所を列挙していきます。
ax.set_xlimで軸の目盛り範囲を指定します。ax.scatter()でs=100, c=zとすることで、プロットのサイズと色(zの値を参考にする)を指定します。ax.view_init()で初期のカメラ位置を指定します。ax.set_xticks([])で目盛りのラベルを消去します。ax.xaxis.set_pane_color((1,1,1,0))で各面を無色にします。ax.xaxis._axinfo["grid"]['color'] = (1,1,1,0)で軸を無色にします。
今回の場合は、立体構造以外の軸や目盛りを消去したいため、上記の設定を行いました。
出力
これにより得られる出力は以下の通りです。
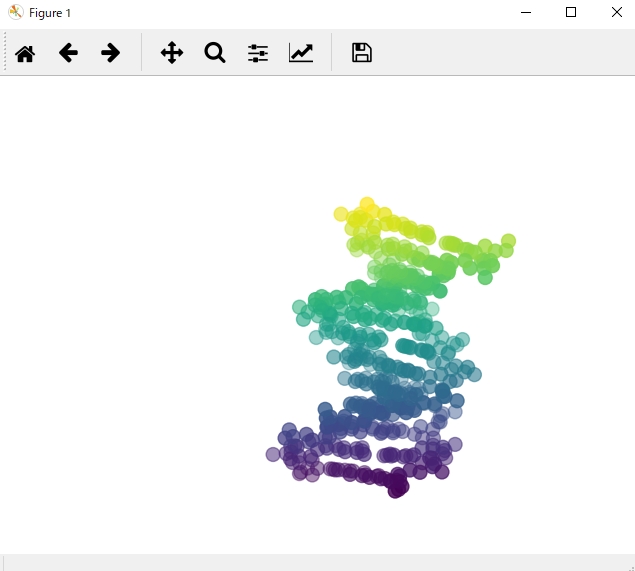
目的の立体データが出力されたことを確認できました。
初期の視点位置の関係でやや中心からずれていますが、視点は手動でも調整できます。